De l’animal à l’Homme, analyse de la diversité génétique des coronavirus. Le projet ANR EPICOREM
Afin de mesurer la présence des coronavirus dans la faune sauvage, les chercheurs ont collecté plus de 4000 prélèvements de mammifères provenant de France, Espagne et Afrique du nord. De nouveaux coronavirus ont été détectés et séquencés, révélant des situations épidémiologiques différentes entre les hôtes.
 Les hérissons étudiés hébergent des Betacoronavirus avec une prévalence de plus de 50%, tandis que les rongeurs hébergent un Betacoronavirus avec une prévalence de 7%, et les lapins de Garenne hébergent des Alphacoronavirus et Betacoronavirus avec une prévalence de 7%. De plus, 14 nouveaux coronavirus (dix Alphacoronavirus et 1 Betacoronavirus) ont été détectés dans 14% des échantillons prélevés chez 10 des 26 espèces de chauves-souris étudiées.
Les hérissons étudiés hébergent des Betacoronavirus avec une prévalence de plus de 50%, tandis que les rongeurs hébergent un Betacoronavirus avec une prévalence de 7%, et les lapins de Garenne hébergent des Alphacoronavirus et Betacoronavirus avec une prévalence de 7%. De plus, 14 nouveaux coronavirus (dix Alphacoronavirus et 1 Betacoronavirus) ont été détectés dans 14% des échantillons prélevés chez 10 des 26 espèces de chauves-souris étudiées.
Cette étude exploratoire souligne la diversité et le dynamisme des coronavirus qui circulent chez la faune sauvage de ces régions, en particulier chez les chauves-souris.
Quelles sont les relations génétiques entre les coronavirus ?
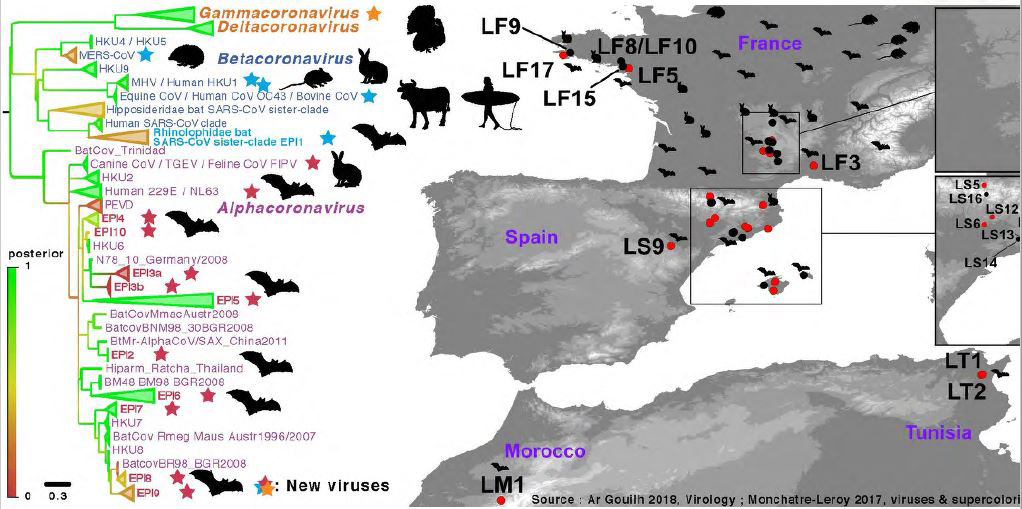
L’équipe a également étudié les liens phylogénétiques entre ces nouvelles espèces virales et d’autres coronavirus identifiés. Des analyses révèlent une parenté pour plusieurs séquences, par exemple six des Alphacoronavirus détectés chez les chauve-souris étudiés ont leur plus proche parent en Europe, et cinq autres sont plus proches de coronavirus décrits en Chine ou à Hongkong.
Y-a-t-il alors une différence entre les coronavirus circulant chez l’animal et les coronavirus humains ? Des travaux menés sur le coronavirus humain OC43 montrent un lien phylogénétique avec le coronavirus bovin (bCoV) ayant émergé il y a un siècle. Ces virus présentent aujourd’hui des combinaisons de mécanismes d’évolution différents (recombinaisons, dérive, sélection) probablement conditionnés par l’écologie de leurs hôtes (Kin, 2015, 2016) et circulent de façon indépendante, illustrant le dynamisme évolutif des coronavirus.
Un modèle naturel pour l’étude des variations génétiques chez l’hôte
En parallèle de ces études sur la macro-diversité des coronavirus, les chercheurs ont suivi une colonie de chauve-souris qui héberge de façon pérenne et par vague saisonnière un Betacoronavirus, groupe-frère du SRAS-CoV (sous genre des Sarbecovirus).
L’objectif ? Analyser les variations sur le génome du virus chez l’hôte pour mieux comprendre la dynamique évolutive du virus. Le génome complet du Betacoronavirus a été analysé par séquençage haut-débit profond sur 18 prélèvements répartis sur 30 mois. Les analyses révèlent des variations de fréquence et des fixations de mutations sur le génome du Betacoronavirus : environ 40 à 60 mutations par an sur le génome du virus, à l’échelle de la colonie suivie. Cette étude met en évidence une évolution génétique rapide de ce Betacoronavirus, avec un taux moyen d’évolution de 1,6.10-3 (soit 0,0016) substitution par site, par an. Ce taux d’évolution rapide contribue à une importante diversité génétique virale.
Grâce à une approche globale « One Health » et multidisciplinaire, le projet EPICOREM a apporté de nouvelles connaissances sur la diversité génétique des coronavirus dans les différents écosystèmes, de la faune sauvage à l’Homme, mais aussi sur les dynamiques d’évolution génétique chez l’hôte. Il a également permis la mise au point de méthodologies d'échantillonnage et de préparation de librairies adaptées aux technologies de nouvelle génération. Ces méthodologies et ces résultats sont essentiels pour mieux comprendre la diversité globale des coronavirus, le passage inter-espèces des coronavirus, le large spectre d’hôtes, et pour mieux caractériser les risques d’émergences à venir.
Les chercheurs poursuivent aujourd’hui leurs travaux sur l’écologie des coronavirus pour mieux documenter l’origine de la diversité virale ainsi que les mécanismes d’évolution et d’adaptation du virus à un autre hôte, et déterminer les sites prioritaires à étudier. Ils intègrent également des dimensions socio-écologiques et anthropologiques dans la construction de modèles d’anticipation et d’estimation des risques d’émergence au sein de projets interdisciplinaires et adoptant la vision One Health selon laquelle les santés animale, humaine et des écosystèmes sont intimement liées.
Les partenaires du projet
Financé par l’Agence nationale de la recherche (ANR), le projet EPICOREM (2014-2018) est coordonné par Astrid Vabret (CHU de Caen) et la direction scientifique est assurée par Meriadeg Le Gouil (Université de Caen Normandie). Il associe l’Institut Pasteur, ainsi que les laboratoires de l’Agence Nationale de Sécurité Sanitaire (ANSES Ploufragan et Nancy), de l’école Vétérinaire d’Alfort (ENVA) et de l'Institut national de recherche pour l’agriculture, l’alimentation et l’environnement (INRAE). Il rassemble des épidémiologistes, des zoologistes, des biologistes moléculaires, des biologistes de l'évolution et des virologues.





